Se usaron las mismas condiciones de PCR para las plantillas sintéticas, excepto que se usó 1 μl de plantilla sintética en 2 ng / μl de ADN de esperma de salmón (Sigma, EE. UU.) En TE en lugar de ADNc. Se utilizó 2x Brilliant III Ultra-Fast QPCR Master Mix (Agilent, EE. UU.)
Las condiciones de ciclismo fueron 95 ° C durante 5-10 min seguidas de 40 ciclos de 95 ° C durante 10-30 segundos y 60 ° C 30-60 segundos. Se realizó un análisis de la curva de fusión (60 ° C a 99 ° C) después del perfil térmico para garantizar la especificidad en la amplificación.
El QPCR de muestras biológicas se realizó en una máquina MX3000P (Stratagene, EE. UU.) Y las reacciones que contienen plantillas sintéticas se realizaron en un Rotorcycler (Qiagen, Alemania). Los cebadores enriquecidos con LNA fueron conjuntos de cebadores de microRNA LNA ™ PCR diseñados por Exiqon (Dinamarca)(1)
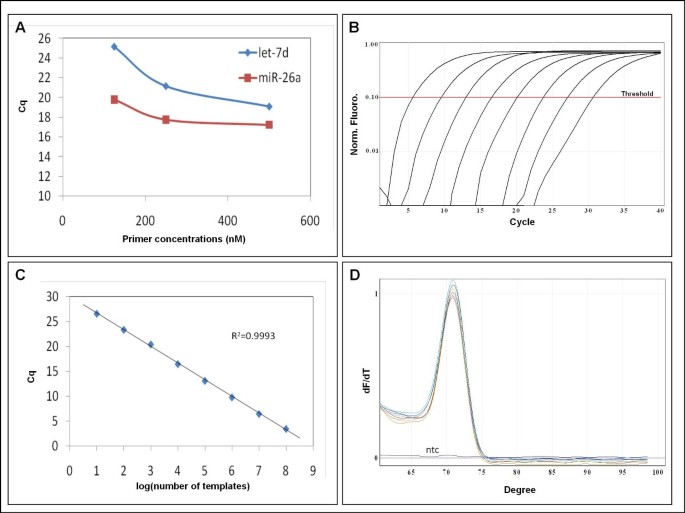
La cuantificación se basó en la determinación del ciclo de cuantificación (Cq) y la eficiencia de la PCR se calculó a partir de la porción logarítmica lineal de las curvas estándar(1)
Referencias Bibliográficas:




